Les lentivirus
Préambule
Je tiens à signaler que le texte ci-dessous constitue une partie de mon rapport bibliographique de DEA (donc ©). Il avait pour thème la plasticité du génome lentiviral.
Plasticité du génome lentiviral et répercussions sur le virus et l’hôte
Sommaire
Introduction
I. La variabilité génétique lentivirale
I. 1. Mécanismes
I. 2. Hétérogénéité de la distribution
de la variabilité génétique
II. Changement de tropisme cellulaire
III. Virulence
IV. Zoonose
V. Réponse immunitaire de l’hôte et échappement
du virus à la neutralisation immune
V. 1. Diversité de la réponse de l’hôte face
à l’infection virale
V. 2. Echappement à la neutralisation immune
Conclusion
Références
Introduction
La famille des Retroviridae est constituée des 7 genres : alpha-,
bêta-, gamma-, delta-, epsilon-rétrovirus, les spumavirus,
et les lentivirus. Le genre lentivirus comprend EIAV (Equine Infectious
Anemia Virus), HIV (Human Immunodeficiency Virus), SIV (Simian Immunodeficiency
Virus), FIV (Feline Immunodeficiency Virus), BIV (Bovine Immunodeficiency
Virus) et SRLV (Small Ruminant Lentiviruses).
Les lentivirus sont responsables de maladies chroniques d’évolution
lente dont les signes cliniques ne se manifestent qu’après
des mois voire des années. Les lentivirus sont des particules
sphériques de 80-100 nm de diamètre dont l’enveloppe
est constituée d’une membrane lipidique cellulaire et des
glycoprotéines virales d’enveloppe.

(Particule d'HIV. Source: www.niaid.nih.gov/daids/ dtpdb/VIRTARG.HTM)
Ils possèdent un génome constitué de deux molécules d’ARN simple brin de polarité positive dont la taille varie de 8 à 10 kb. Les génomes rétroviraux sont constitués de 3 gènes de structure. Le gène gag code les protéines de la capside, de la nucléocapside et de la matrice. Le gène pol code la protéase, la transcriptase inverse (RT pour Reverse Transcriptase), l’intégrase et une protéine supplémentaire, la désoxyuridine triphosphatase ou dUTPase, chez les lentivirus des Ongulés. Le gène env code la glycoprotéine d’enveloppe de surface (SU) et la glycoprotéine d’enveloppe transmembranaire (TM). Outre ces gènes communs à tous les rétrovirus, les lentivirus contiennent des cadres ouverts de lecture supplémentaires codant des petites protéines avec des fonctions accessoires ou régulatrices.

(Génome d'HIV. Source: http://biology.kenyon.edu/courses/biol63/chime2000/HIV-1/FRAMES/genome.html)
Les lentivirus peuvent être divisés en deux groupes sur la base de leur tropisme cellulaire : EIAV, CAEV et MVV infectent la lignée monocyte-macrophage, alors que HIV, SIV et FIV infectent, en plus des macrophages, les lymphocytes T portant l’antigène de surface de la classe de différenciation 4 (CD4+). Les sites primaires de réplication virale comprennent la moelle osseuse et les organes lymphoïdes secondaires dont la rate et les nœuds lymphatiques. Ces tissus constituent le réservoir des monocytes et lymphocytes infectés qui pourront être distribués vers les autres organes où les monocytes maturent en macrophages et les lymphocytes sont activés. La différenciation et/ou l’activation de ces cellules induit l’expression des gènes viraux et la production de virus. Cette différenciation et/ou activation peut se faire dans le sang ou les tissus périphériques.
La première étape du cycle viral est
l’interaction du virus avec la cellule cible par la fixation de
la glycoprotéine de surface au récepteur cellulaire. Parmi
les récepteurs cellulaires, sont distingués deux types
de récepteurs à chémokines CCR5 et CXCR4 : ce sont
des protéines à sept passages transmembranaires couplées
à des protéines G. Les chémokines forment une classe
de cytokines proinflammatoires chimiotactiques capables de recruter
des leucocytes vers le site inflammatoire. Elles sont définies
par un motif structural commun composé de quatre cystéines.
Chez les bêta-chémokines ou CC chémokines, les deux
premières cystéines sont adjacentes, alors que chez les
alpha-chémokines ou CXC chémokines, les deux cystéines
sont séparées par un acide aminé. L’entrée
du virus se fait par fusion de l’enveloppe virale avec la membrane
cellulaire. Après une étape de décapsidation, le
génome lentiviral est rétrotranscrit par la RT virale
en ADN double brin dans le cytoplasme. Cet ADN double brin est transporté
vers le noyau cellulaire et s’intègre aléatoirement
dans le génome cellulaire. Une fois intégré, l’ADN
double brin prend alors le nom de provirus. C’est à ce
moment que peut intervenir une phase de latence : certaines cellules
sont permissives pour les étapes précoces du cycle de
vie du virus mais ne permettent pas l’expression des gènes
viraux qui doit être induite par des facteurs de transcription
transitoirement activés (Persaud et al. 2003. J Virol).
La machinerie cellulaire de transcription et de traduction est ensuite
utilisée pour permettre l’expression du génome proviral.
Les ARN sont transportés vers le cytoplasme où le génome
ARN est encapsidé et les autres transcrits codent les protéines
de structure et les enzymes virales nécessaires au cycle viral.
La particule virale est enveloppée lors de la libération
de virus par la cellule infectée.
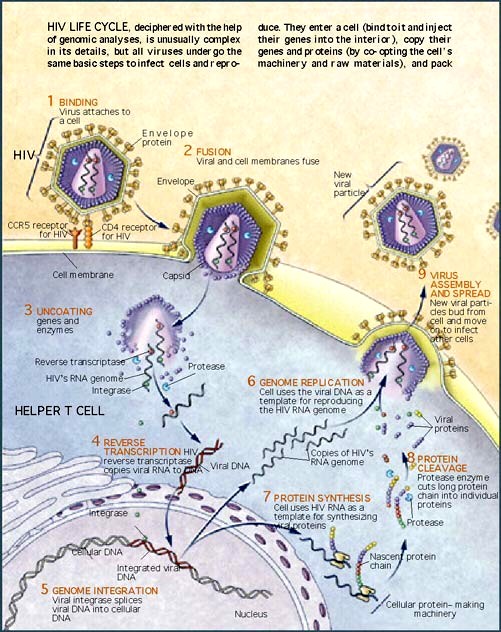
(Cycle Viral d'HIV. Source: http://hiv.buffalo.edu//hivlifecycle.shtml)
OU de façon animée ce même cycle : http://hsc.virginia.edu/med-ed/micro/images/vir10/1.swf
Dommage mais la suite est ma propriété
intellectuelle ©.
Mais vous pouvez me contacter
pour des renseignements!
Lecture
complémentaire:
Article
de La Recherche de décembre 2003.